层次聚类和Kmeans
(1) 计算两两样本之间的距离;
(2) 将距离最小的两个类合并成一个新类;
(3) 重新计算新类与所有类之间的距离;
(4) 重复(2)、(3),直到所有类最后合并成一类。
层次聚类例子
import scipy
import scipy.cluster.hierarchy as sch
from scipy.cluster.vq import vq,kmeans,whiten
import numpy as np
import matplotlib.pylab as plt
#生成待聚类的数据点,这里生成了20个点,每个点4维:
points=scipy.randn(20,4)
#加一个标签进行区分
A=[chr(i+ord('A')) for i in range(20)]
#层次聚类
#生成点与点之间的距离矩阵,这里用的欧氏距离:
disMat = sch.distance.pdist(points,'euclidean')
#进行层次聚类,method='average'用平均数来计算新合成类别的坐标:
Z=sch.linkage(disMat,method='average')
#将层级聚类结果以树状图表示出来并保存为plot_dendrogram.png
P=sch.dendrogram(Z,labels=A)
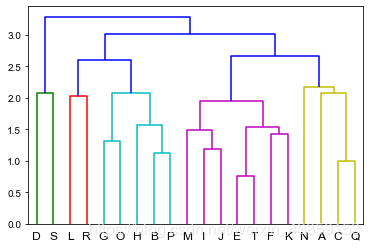
优点
1、距离和规则的相似度容易定义,限制少;
2、不需要预先制定聚类数;
3、可以发现类的层次关系。
缺点
1、计算复杂度太高。
(1) 首先确定一个k值;
(2) 随机给定k个质心;
(3) 对数据集中每一个点,计算其与每一个质心的距离(如欧式距离),离哪个质心近,就划分到那个质心所属的集合;
(4) 把所有数据归好集合后,一共有k个集合。然后重新计算每个集合的新的质心;
(5) 重复(3) (4) ;
(6) 如果新计算出来的质心和原来的质心之间的距离小于某一个设置的阈值,算法终止。
def Kmeans_analysis(X,k,train=True):
y_pred=None
if train==True:
if X.shape[1]==2:
silhouette = []
sse = []
for n_clusters in k:
n_clusters = n_clusters
fig, (ax1, ax2) = plt.subplots(1, 2)
fig.set_size_inches(18, 7)
ax1.set_xlim([-0.1, 1])
ax1.set_ylim([0, X.shape[0] + (n_clusters + 1) * 10])
clusterer = KMeans(n_clusters=n_clusters, random_state=10).fit(X)
cluster_labels = clusterer.labels_
silhouette_avg = silhouette_score(X, cluster_labels)
silhouette.append(silhouette_avg)#计算轮廓系数
#meanDispersions.append(sum(np.min(cdist(X, clusterer.cluster_centers_, 'euclidean'), axis=1)) / X.shape[0])#计算平均畸变程度,跟SSE同一原理
sse.append(clusterer.inertia_)#计算SSE
print("当 k =", n_clusters,"平均轮廓系数为:", silhouette_avg)
sample_silhouette_values = silhouette_samples(X, cluster_labels)
y_lower = 10
for i in range(n_clusters):
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i)/n_clusters)
ax1.fill_betweenx(np.arange(y_lower, y_upper)
,ith_cluster_silhouette_values
,facecolor=color
,alpha=0.7)
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10
ax1.set_title("轮廓系数图")
ax1.set_xlabel("轮廓系数")
ax1.set_ylabel("聚类标签")
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([])
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters)
ax2.scatter(X[:, 0], X[:, 1],marker='o',s=8,c=colors)
centers = clusterer.cluster_centers_
ax2.scatter(centers[:, 0], centers[:, 1], marker='x',c="red", alpha=1, s=200)#画质心点
ax2.set_title("数据可视化")
ax2.set_xlabel("x1")
ax2.set_ylabel("x2")
plt.suptitle("当k={}时,Kmeans轮廓系数图".format(n_clusters),fontsize=14, fontweight='bold')
fig, (ax2, ax3) = plt.subplots(1, 2,figsize=(9,4))
ax2.plot(k,silhouette, 'bo-', color='k')
ax2.grid()
ax2.set_title("轮廓系数确定k值")
ax2.set_xlabel("k值")
ax2.set_ylabel("轮廓系数")
ax3.plot(k, sse, 'bo-', color='k')
ax3.grid()
ax3.set_title('肘部法确定k值')
ax3.set_xlabel("k值")
ax3.set_ylabel('SSE')
else:
silhouette = []
sse = []
for n_clusters in k:
n_clusters = n_clusters
fig, ax1 = plt.subplots(1,1,figsize=(9,7))
ax1.set_xlim([-0.1, 1])
ax1.set_ylim([0, X.shape[0] + (n_clusters + 1) * 10])
clusterer = KMeans(n_clusters=n_clusters, random_state=10).fit(X)
cluster_labels = clusterer.labels_
silhouette_avg = silhouette_score(X, cluster_labels)
silhouette.append(silhouette_avg)#计算轮廓系数
sse.append(clusterer.inertia_)#计算SSE
#meanDispersions.append(sum(np.min(cdist(X, clusterer.cluster_centers_, 'euclidean'), axis=1)) / X.shape[0])#计算平均畸变程度,跟SSE同一原理
print("当 k =", n_clusters,"平均轮廓系数为:", silhouette_avg)
sample_silhouette_values = silhouette_samples(X, cluster_labels)
y_lower = 10
for i in range(n_clusters):
ith_cluster_silhouette_values = sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i)/n_clusters)#颜色设置
ax1.fill_betweenx(np.arange(y_lower, y_upper)
,ith_cluster_silhouette_values
,facecolor=color
,alpha=0.7)
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
y_lower = y_upper + 10
ax1.set_title("当k={}时,Kmeans轮廓系数图".format(n_clusters))
ax1.set_xlabel("轮廓系数")
ax1.set_ylabel("聚类标签")
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([])
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
fig, (ax2, ax3) = plt.subplots(1, 2,figsize=(9,4))
ax2.plot(k,silhouette, 'bo-', color='k')
ax2.grid()
ax2.set_title("轮廓系数确定k值")
ax2.set_xlabel("k值")
ax2.set_ylabel("轮廓系数")
ax3.plot(k, sse, 'bo-', color='k')
ax3.grid()
ax3.set_title('肘部法确定k值')
ax3.set_xlabel("k值")
ax3.set_ylabel('SSE')
else:
cluster=KMeans(n_clusters=k,random_state=0).fit(X)
centroid=cluster.cluster_centers_#质心点
y_pred=cluster.labels_#预测
print('质心点:',centroid)
return y_pred
k值选择问题
轮廓系数定义:
s=disMeanout−disMeaninmax(disMeanout,disMeanin)
s=\frac{d i s M e a n_{o u t}-d i s M e a n_{i n}}{m a x\left(d i s M e a n_{o u t}, d i s M e a n_{i n}\right)}
s=max(disMeanout,disMeanin)disMeanout−disMeanin
其中,disMeanintd i s M e a n_{int}disMeanint为该点与本类其他点的平均距离,disMeanoutd i s M e a n_{o u t}disMeanout为该点与非本类点的平均距离。该值取值范围为[−1,1][−1,1][−1,1], 越接近1则说明分类越优秀。在sklearn中函数silhouette_score()计算所有点的平均轮廓系数,而
silhouette_samples()返回每个点的轮廓系数。
SSE:
随着聚类数k的增大,样本划分会更加精细,每个簇的聚合程度会逐渐提高,那么误差平方和SSE自然会逐渐变小。
当k小于真实聚类数时,由于k的增大会大幅增加每个簇的聚合程度,故SSE的下降幅度会很大,而当k到达真实聚类数时,再增加k所得到的聚合程度回报会迅速变小,所以SSE的下降幅度会骤减,然后随着k值的继续增大而趋于平缓,也就是说SSE和k的关系图是一个手肘的形状,而这个肘部对应的k值就是数据的真实聚类数
import numpy as np
import matplotlib.cm as cm
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
from scipy.spatial.distance import cdist
from sklearn.metrics import silhouette_score
from sklearn.metrics import silhouette_samples
plt.rcParams[u'font.sans-serif'] = ['Arial Unicode MS']
X, y = make_blobs(n_samples=500,n_features=3,centers=4,random_state=100)
plt.scatter(X[:,0],X[:,1],marker='o',s=15)
cluster=KMeans(n_clusters=4,random_state=0).fit(X)
centroid=cluster.cluster_centers_#质心点
cluster.inertia_#查看总距离平方和,SSE
y_pred=cluster.labels_#预测
colors = ["red","pink","orange","gray"]
for i in range(4):
plt.scatter(X[y_pred==i,0],X[y_pred==i,1],marker='o',s=15,color=colors[i])
plt.scatter(centroid[:,0],centroid[:,1],marker='x',color='k')
#轮廓系数判断分几类
silhouette_score(X,y_pred)#轮廓系数,越靠近1越好
silhouette_samples(X,y_pred)#每一个样本轮廓系数,所有样本的轮廓系数之和再取平均为轮廓系数
Kmeans_analysis(X,k=[2,3,4],train=True)
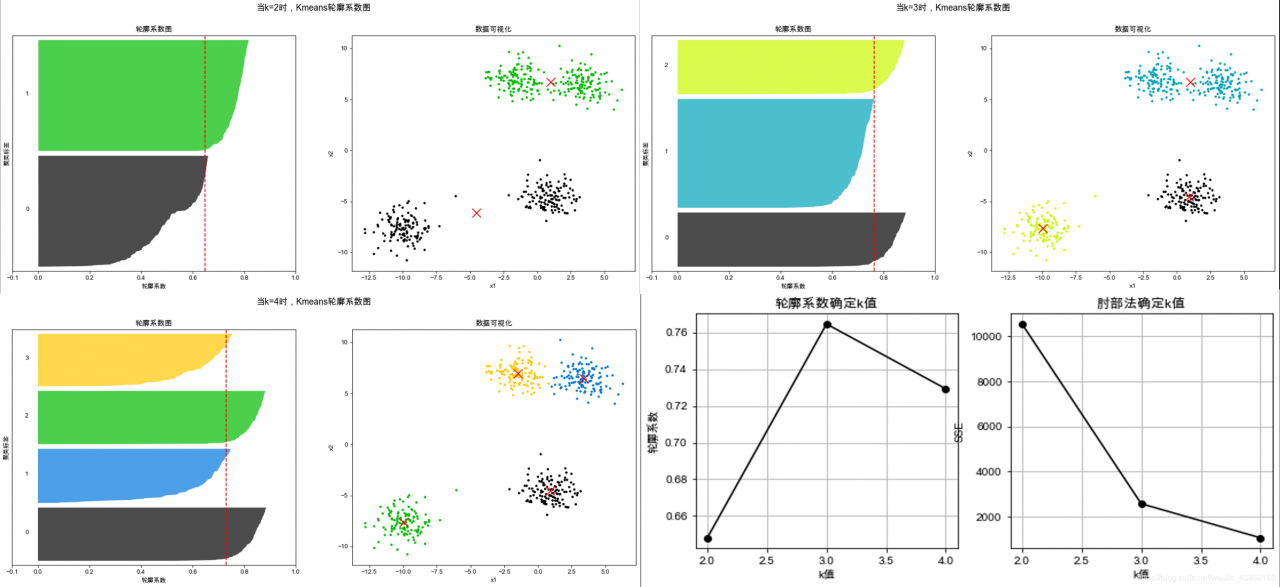
y_pred=Kmeans_analysis(X,k=3,train=False)#最后选择将其分为3类
K-Means的优缺点
优点:
1、原理比较简单,实现也是很容易,收敛速度快。
2、当结果簇是密集的,而簇与簇之间区别明显时, 它的效果较好。
3、主要需要调参的参数仅仅是簇数k。
缺点:
1、K值需要预先给定,很多情况下K值的估计是非常困难的。
2、K-Means算法对初始选取的质心点是敏感的,不同的随机种子点得到的聚类结果完全不同 ,对结果影响很大。
3、对噪音和异常点比较的敏感。
作者:S_ssssssk