Python——Pandas保存数据为HDF5格式时应注意数据类型
在平常的数据存取过程中,我们常常使用csv格式的文件,此格式的文件具有可直接打开、直接编辑等等优点,且使用Python读取csv格式的文件的速度比读取txt格式的更快。由于最近常常需要反复处理几个百万行乃至千万行的数据文件,所以即使我使用了csv格式的文件,读取速度也有显得有些慢,秉持着着“节约时间就等于延长生命”的精神,我四处寻找能够进一步减少从文件中读取数据的时间的方法。这时,我看到了文章: Pandas 中 read_csv 与 read_hdf 速度对比,于是决定着手尝试使用HDF5格式,但是,最初的结果显示使用HDF5格式的文件提升的效率没有链接中的文章给出的那么多,且使用csv格式保存的数据占用的存储空间要大于原先的csv格式的文件(见后文中的图)。通过观察,我发现了问题的症结所在——数据类型。在读取csv并生成pd.DataFrame时以及将数据保存到HDF5文件中时,数据类型是Object(可以理解为字符串),如果使用astype()方法将数据类型转换为numpy.float32,则可以缩小生成的HDF5格式的文件的体积,也可以提高后续读取此文件的速度。
据我推测数据类型转换能够减小文件体积及提高后续读取速度的原因有二:
1.在绝大多数情况下,字符串占用的存储空间比浮点数占用的大(小部分情况之一:字符串是一个小的整数),因此转换数据类型之后可以减少文件占用的硬盘空间;
2.文件体积的缩小以及明确的数据类型减少了硬盘I/O过程占用的时间(这也是读取数据过程中最耗费时间的一个过程)以及类型转换的时间。
结论已经在上面说完了,下文是我自己做的简单读取时间对比测试的过程(可以略过不看):
一、文件准备
我使用原始的数据是某模拟软件生成的格式比较规整的txt格式的文件,大致内容如下图:

我使用简单的Shell脚本来将它转换成了csv格式的文件:
#!/bin/bash
#将原始文件转换为格式为csv格式的文件
cat $1.txt | tr "/[ ]/" "," > $2.csv
#参数1是需要被转换txt文件的名称
#参数2是最终产生的csv的文件名称
生成的csv格式的文件信息如下:
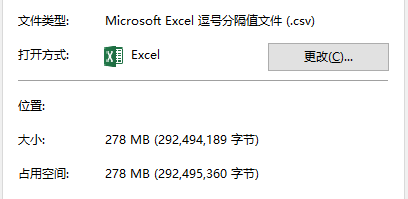
二、读取csv格式的文件中的数据保存到HDF5格式的文件中
直接使用导入python自带的csv模块,然后使用csv.reader方法将文件中的数据读入,然后将数据转换为DataFrame,随后使用pandas的to_hdf()方法将数据存储到HDF5格式的文件中。
import csv
import os
import numpy as np
import pandas as pd
csvfilename='Result.csv'
filename='datahdf5.h5'
csvfile=open(csvfilename)
reader=csv.reader(csvfile)
#不进行类型转换
df = pd.DataFrame(reader)
#将数据类型指定为astype(np.float32)
#df = pd.DataFrame(reader).astype(np.float32)
df.to_hdf(filename, 'a')
这里需要注意:直接读取整个文件的方式将会占用大量的内存,如果文件过大,甚至可能导致memory error出现。
下面是使用Object类型存储数据和使用numpy.float32类型存储数据时生成的HDF5格式的文件的大小信息:
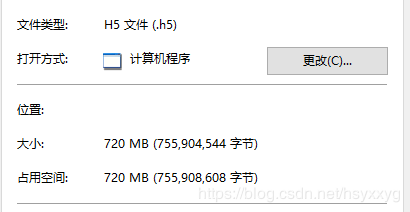
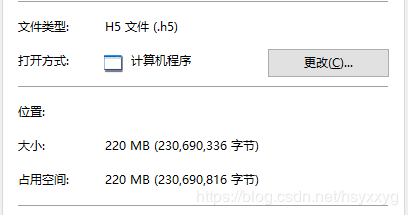
前面提到过,除了文件尺寸的大幅缩减,在读取速度方面,使用numpy.float32类型存储数据也具有极大的优势,下面是我直接读取以上两个文件然后将数据保存到一个pandas.DataFrame中使用的时间的对比,使用的代码如下:
#读取.h5文件,将数据保存到一个DataFrame中,并打印数据类型。
import pandas as pd
data = pd.read_hdf("datahdf5.h5")
df = pd.DataFrame(data)
print(df.dtypes)
下面是数据和输出的数据类型结果,需要说明的是:1.测试读取数据的时间时,我使用的是cygwin配合time命令,而非原生的类Unix系统上的命令;2.具体的时间结果只代表我个人的计算机上的结果,不同的计算机上耗费的的时间可能不同,但是时间的增减趋势应该是相同的;3.更加严谨的方式是测量多次,然后求一个均值,但是因为我只是想说明这种趋势,所以没有进行更加严谨的测试。
结果如下:
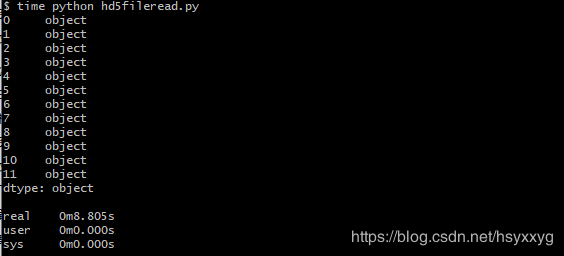
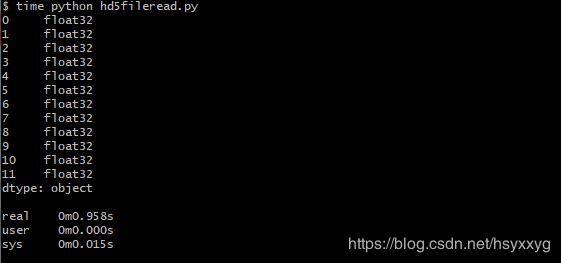 通过上面的对比,我们可以看出:使用pandas将数据存储为HDF5格式的文件时,根据数据的特点指定数据类型,而非直接使用Object类型保存数据,我们就可以缩小HDF5格式的文件占用的空间,同时减小了再次读取文件中的数据时消耗的时间。
通过上面的对比,我们可以看出:使用pandas将数据存储为HDF5格式的文件时,根据数据的特点指定数据类型,而非直接使用Object类型保存数据,我们就可以缩小HDF5格式的文件占用的空间,同时减小了再次读取文件中的数据时消耗的时间。
作者:宋体的微软雅黑