python 如何通过KNN来填充缺失值
# 加载库
import numpy as np
from fancyimpute import KNN
from sklearn.preprocessing import StandardScaler
from sklearn.datasets import make_blobs
# 创建模拟特征矩阵
features, _ = make_blobs(n_samples = 1000,
n_features = 2,
random_state = 1)
# 标准化特征
scaler = StandardScaler()
standardized_features = scaler.fit_transform(features)
standardized_features
# 制造缺失值
true_value = standardized_features[0,0]
standardized_features[0,0] = np.nan
standardized_features
# 预测
features_knn_imputed = KNN(k=5, verbose=0).fit_transform(standardized_features)
# features_knn_imputed = KNN(k=5, verbose=0).complete(standardized_features)
features_knn_imputed
# #对比真实值和预测值
print("真实值:", true_value)
print("预测值:", features_knn_imputed[0,0])
# 加载库
import numpy as np
from fancyimpute import KNN
from sklearn.preprocessing import StandardScaler
from sklearn.datasets import make_blobs
# 创建模拟特征矩阵
features, _ = make_blobs(n_samples = 1000,
n_features = 2,
random_state = 1)
# 标准化特征
scaler = StandardScaler()
standardized_features = scaler.fit_transform(features)
standardized_features
# 制造缺失值
true_value = standardized_features[0,0]
standardized_features[0,0] = np.nan
standardized_features
# 预测
features_knn_imputed = KNN(k=5, verbose=0).fit_transform(standardized_features)
# features_knn_imputed = KNN(k=5, verbose=0).complete(standardized_features)
features_knn_imputed
# #对比真实值和预测值
print("真实值:", true_value)
print("预测值:", features_knn_imputed[0,0])
真实值: 0.8730186113995938
预测值: 1.0955332713113226
补充:scikit-learn中一种便捷可靠的缺失值填充方法:KNNImputer
在数据挖掘工作中,处理样本中的缺失值是必不可少的一步。其中对于缺失值插补方法的选择至关重要,因为它会对最后模型拟合的效果产生重要影响。
在2019年底,scikit-learn发布了0.22版本,此次版本除了修复之前的一些bug外,还更新了很多新功能,对于数据挖掘人员来说更加好用了。其中我发现了一个新增的非常好用的缺失值插补方法:KNNImputer。这个基于KNN算法的新方法使得我们现在可以更便捷地处理缺失值,并且与直接用均值、中位数相比更为可靠。利用“近朱者赤”的KNN算法原理,这种插补方法借助其他特征的分布来对目标特征进行缺失值填充。
下面,就让我们用实际例子来看看KNNImputer是如何使用的吧使用KNNImputer需要从scikit-learn中导入:
from sklearn.impute import KNNImputer
先来一个小例子开开胃,data中第二个样本存在缺失值。
data = [[2, 4, 8], [3, np.nan, 7], [5, 8, 3], [4, 3, 8]]
KNNImputer中的超参数与KNN算法一样,n_neighbors为选择“邻居”样本的个数,先试试n_neighbors=1。
imputer = KNNImputer(n_neighbors=1)
imputer.fit_transform(data)

可以看到,因为第二个样本的第一列特征3和第三列特征7,与第一行样本的第一列特征2和第三列特征8的欧氏距离最近,所以缺失值按照第一个样本来填充,填充值为4。那么n_neighbors=2呢?
imputer = KNNImputer(n_neighbors=2)
imputer.fit_transform(data)

此时根据欧氏距离算出最近相邻的是第一行样本与第四行样本,此时的填充值就是这两个样本第二列特征4和3的均值:3.5。
接下来让我们看一个实际案例,该数据集来自Kaggle皮马人糖尿病预测的分类赛题,其中有不少缺失值,我们试试用KNNImputer进行插补。
import numpy as np
import pandas as pd
import pandas_profiling as pp
import matplotlib.pyplot as plt
import seaborn as sns
sns.set(context="notebook", style="darkgrid")
import warnings
warnings.filterwarnings('ignore')
%matplotlib inline
from sklearn.impute import KNNImputer
#Loading the dataset
diabetes_data = pd.read_csv('pima-indians-diabetes.csv')
diabetes_data.columns = ['Pregnancies', 'Glucose', 'BloodPressure', 'SkinThickness',
'Insulin', 'BMI', 'DiabetesPedigreeFunction', 'Age', 'Outcome']
diabetes_data.head()
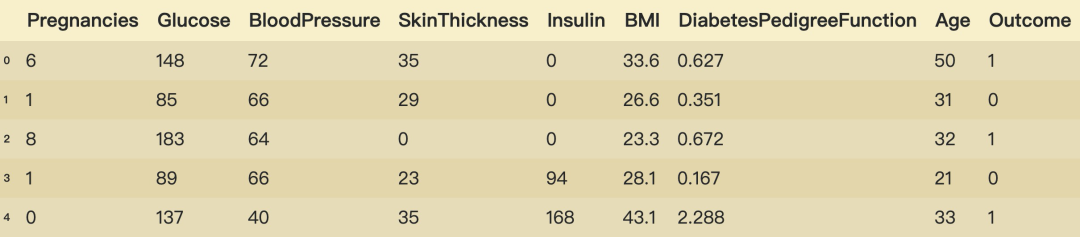
在这个数据集中,0值代表的就是缺失值,所以我们需要先将0转化为nan值然后进行缺失值处理。
diabetes_data_copy = diabetes_data.copy(deep=True)
diabetes_data_copy[['Glucose','BloodPressure','SkinThickness','Insulin','BMI']] = diabetes_data_copy[['Glucose','BloodPressure','SkinThickness','Insulin','BMI']].replace(0, np.NaN)
print(diabetes_data_copy.isnull().sum())
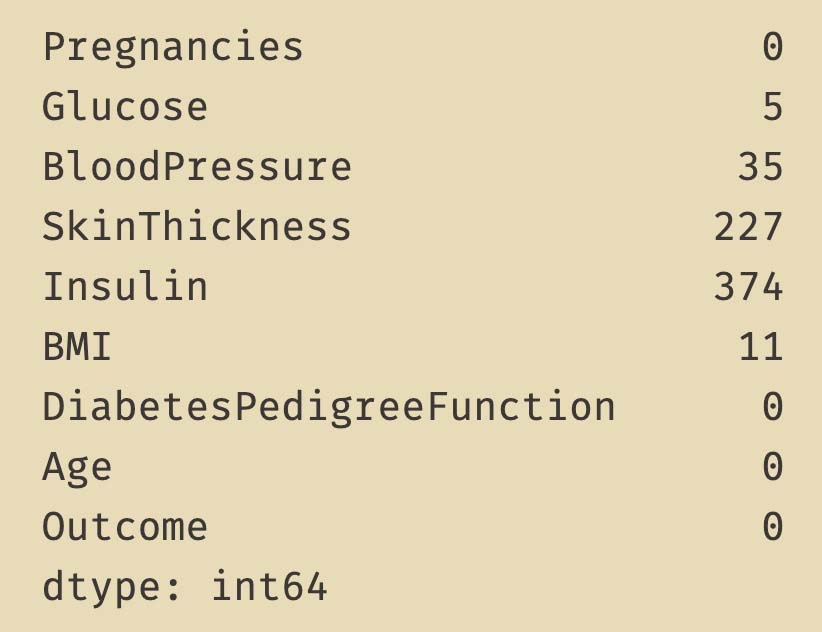
在本文中,我们尝试用DiabetesPedigreeFunction与Age,对BloodPressure中的35个缺失值进行KNNImputer插补。
先来看一下缺失值都在哪几个样本:
null_index = diabetes_data_copy.loc[diabetes_data_copy['BloodPressure'].isnull(), :].index
null_index

imputer = KNNImputer(n_neighbors=10)
diabetes_data_copy[['BloodPressure', 'DiabetesPedigreeFunction', 'Age']] = imputer.fit_transform(diabetes_data_copy[['BloodPressure', 'DiabetesPedigreeFunction', 'Age']])
print(diabetes_data_copy.isnull().sum())
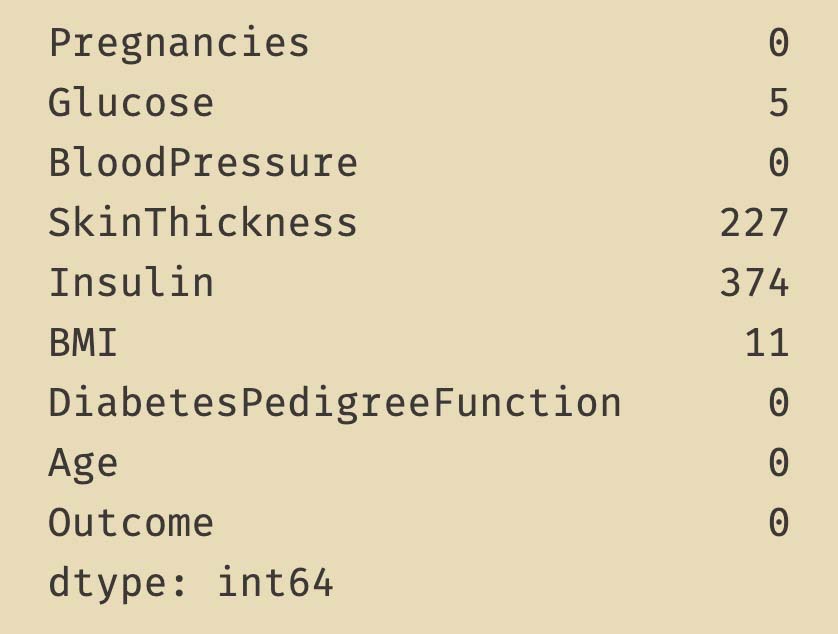
可以看到现在BloodPressure中的35个缺失值消失了。我们看看具体填充后的数据(只截图了部分):
diabetes_data_copy.iloc[null_index]
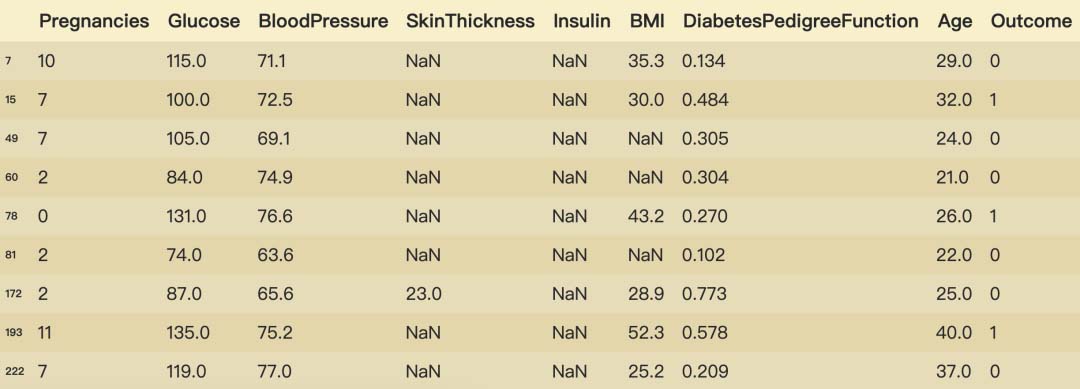
到此,BloodPressure中的缺失值已经根据DiabetesPedigreeFunction与Age运用KNNImputer填充完成了。注意的是,对于非数值型特征需要先转换为数值型特征再进行KNNImputer填充操作,因为目前KNNImputer方法只支持数值型特征(ʘ̆ωʘ̥̆‖)՞。