机器学习入门 --- K-means、DBSCAN聚类算法(概念、图解、代码示例)
聚类是把相似的东西分到一组,它是一个无监督问题,没有标签使用
难点:
对于有标签的有监督学习问题,标签可以便于我们来评估模型,无监督学习问题在评估上比较难一点
对于不同的参数组合,得到的学习结果,因为比较难对模型做评估,所以不能通过一个精确度的好坏来选择参数组合
K-MEANS算法是聚类问题中,最简单,也是最实用的一个算法
基本概念一个数据放进来,需要指定K值,来声明要得到簇的个数
质心:一个簇的数据均值,即向量各维取平均即可(迭代时使用)
距离的度量:常用欧几里得距离和余弦相似度(数据需先标准化)
优化目标通过目标函数进行不断地优化、求解
min∑i=1K∑x∈Cidist(ci,x)2min\sum_{i=1}^{K}\sum_{x \in C_i}dist(c_i,x)^2mini=1∑Kx∈Ci∑dist(ci,x)2
∑i=1K\sum_{i=1}^{K}∑i=1K 表示一共有多少个簇,对 KKK 个簇优化
∑x∈Cidist(ci,x)2\sum_{x \in C_i}dist(c_i,x)^2∑x∈Cidist(ci,x)2 表示每个簇的值到其中心点的距离
工作流程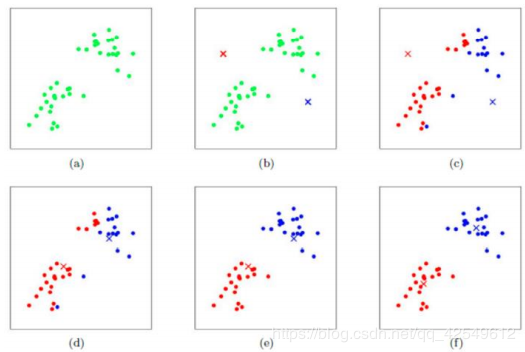
对于以上这个例子,我们设置 K=2K = 2K=2 ,首先要初始化质心(随机),如 bbb 图所示,通过计算数据中所有的点到分别到这两个质心的距离,哪个距离短,就归于哪一簇,c图为初步结果,划分出了红色、蓝色的两簇,但效果不是很好,需要进一步优化,接下来根据所有的红、蓝点,重新计算质心,进行质心的更新,得到 ddd 图所示质心位置,更新质心后,再次迭代,计算所有的点到分别到这两个质心的距离,哪个距离短,就归于哪一簇,则得到 eee 图的结果,再次更新质心,进行迭代… 直到所有样本点都不再发生变化了,更新基本结束了
优势:
算法的理解、操作简单 使用快速,适合常规数据集劣势:
对于一个新的样本,没有标签很难知道要去分多少个簇,K值难确定,通常情况下要设置多组参数实验看效果
质点的初始值影响大
复杂度与样本呈线性关系,数据量庞大时,计算量会很大
因为数据的簇是不确定的,很难发现任意形状的簇,聚类效果不是很好。
比如下图这种,K-means聚类算法会根据初始化质心划分成这样四个簇,但实际上应该是里面三个簇,外面再环绕着一个簇。

https://www.naftaliharris.com/blog/visualizing-k-means-clustering/
DBSCAN算法基本概念:(Density-Based Spatial Clustering of Applications with Noise)
基于密度的噪声应用空间聚类
各点描述:
假设 A 为核心对象,画了一个圈,圈中了 A′、A′′、A′′′A'、A''、A'''A′、A′′、A′′′ 三个点,并以 A′、A′′、A′′′A'、A''、A'''A′、A′′、A′′′ 为核心对象继续向外扩展,最终到了 B、CB、CB、C 两点时无法再进行扩展,即 B、CB、CB、C 两点为边界点,NNN 点没有一个圈能涉及到它,它不属于任何一个类簇的点,是离群点
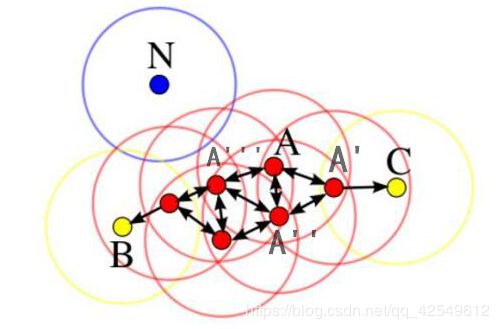
因为此算法可检测离群点,所以说 DBSCAN算法还可以做异常检测等任务
三个参数:
参数D:输入数据集 参数ϵ:指定半径 MinPts:密度阈值流程:
1. 标记所有对象为 unvisited(初始时,所有数据都是未访问的)
2. Do
3. 随机选择-一个 uvisited 对象 p
4. 标记 p 为 visited
5. if p 的 ϵ- 领域至少有 MinPts 个对象
6. 创建一个新族 C, 并把 p 添加到 C
7. 令 N 为 p 的 ϵ- 领域中的对象集合
8. For N 中每个点 p'
9. if p' 是 umvisited
10. 标记 p' 为 visited
11. if p' 的 ϵ- 领域至少有 MinPs 个对象,把这些对象添加到 N
12. if p' 还不是任何簇的成员,把 p' 添加到C
13. End for
14. 输出C:
15. Else 标记p为噪声;
16. Until 没有标记为uwvisited的对象:
参数选择:
半径 ϵ:可以根据 K 距离来设定:找突变点K距离:给定数据集P={p(i); i=0,1,…n},计算点P(i)到集合D的子集S中所有点之间的距离,距离按照从小到大的顺序排序,d(k) 就被称为 K距离,d(k) 集中数据突变的位置即为突变点,根据这里来设定半径 ϵ MinPts: K距离中 K 的值,一般取的小一些,多次尝试
优势:
不需要指定簇个数 擅长找到离群点(检测任务) 可以发现任意形状的簇 两个参数就够了劣势:
高维数据有些困难(可能会报内存溢出,可以做降维) Sklearn中效率很慢(数据削减策略) 参数难以选择(参数对结果的影响非常大)相比于 K-MEANS算法,DBSCAN算法可以很好的把这个笑脸进行份簇
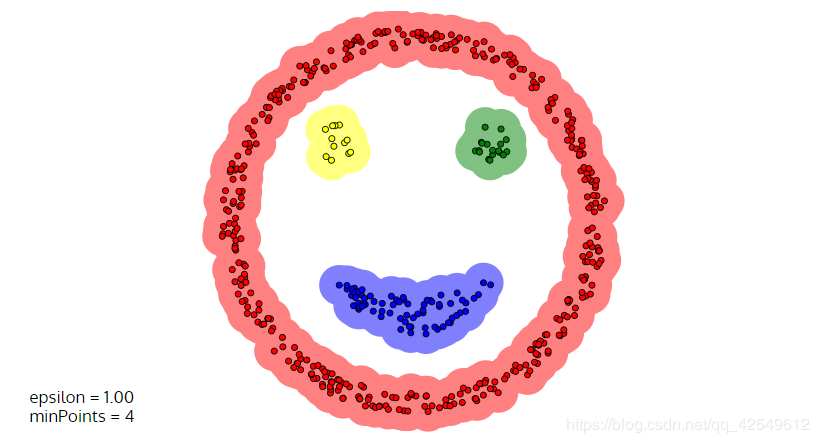
https://www.naftaliharris.com/blog/visualizing-dbscan-clustering/
聚类实践在这个里使用一个啤酒相关的数据进行聚类演示
# beer dataset
import pandas as pd
beer = pd.read_csv('data.txt', sep=' ')
beer.head()
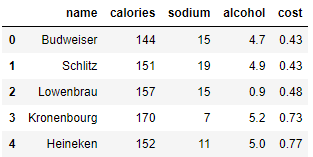
构建聚类的数据输入
X = beer[["calories","sodium","alcohol","cost"]]
K-means 算法
from sklearn.cluster import KMeans
# 使用3个簇做聚类
km = KMeans(n_clusters=3).fit(X)
# 使用2个簇做聚类
km2 = KMeans(n_clusters=2).fit(X)
# 将聚类结果整合到数据中
beer['cluster'] = km.labels_
beer['cluster2'] = km2.labels_
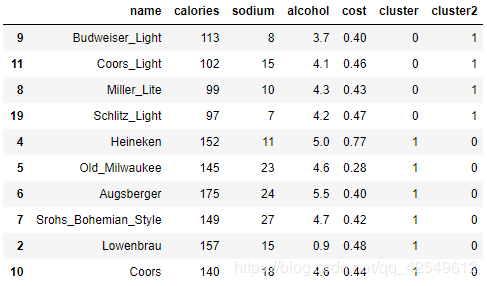
# 根据 cluster 排序后展示
beer.sort_values('cluster').head(10)
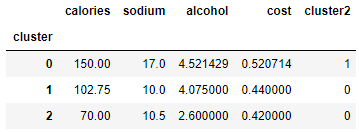
基于每个簇,查看各项平均值:
beer.groupby("cluster").mean()
beer.groupby("cluster2").mean()
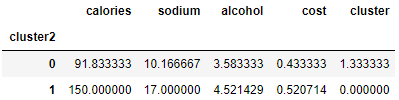
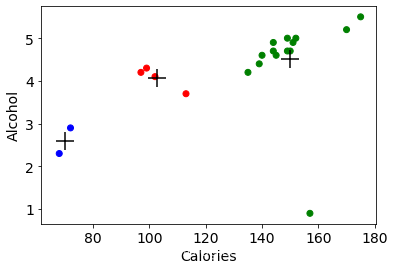
通过图像看一下3簇的聚类情况
import numpy as np
import matplotlib.pyplot as plt
# 拿到中心点
centers = beer.groupby("cluster").mean().reset_index()
# 大小
plt.rcParams['font.size'] = 14
# 颜色
colors = np.array(['red', 'green', 'blue', 'yellow'])
# 画数据点
plt.scatter(beer["calories"], beer["alcohol"],c=colors[beer["cluster"]])
# 画中心点
plt.scatter(centers.calories, centers.alcohol, linewidths=3, marker='+', s=300, c='black')
plt.xlabel("Calories")
plt.ylabel("Alcohol")
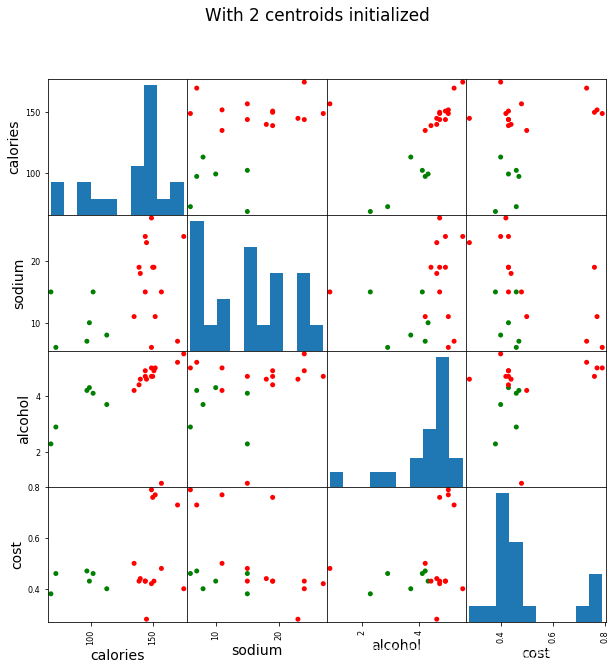
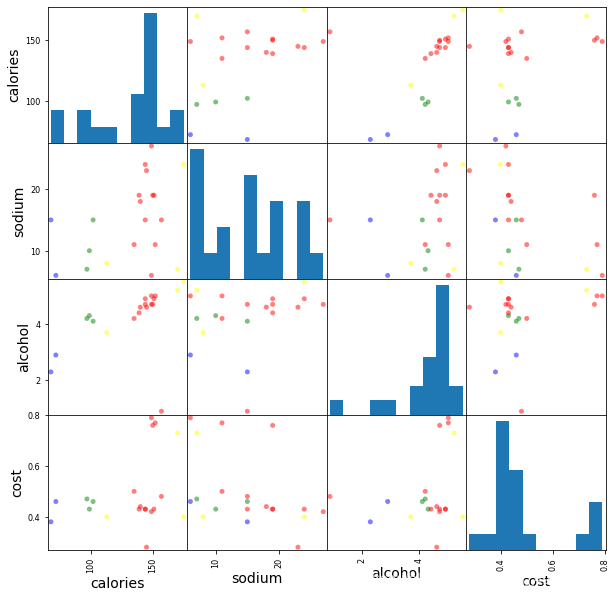
这张图展示出的只是在 Calories 和 Alcohol 两个维度上的关系,但我们的数据是4个维度,下面再看一下这4个维度的两两关系
# 2簇
scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster2"]], figsize=(10,10))
plt.suptitle("With 3 centroids initialized")
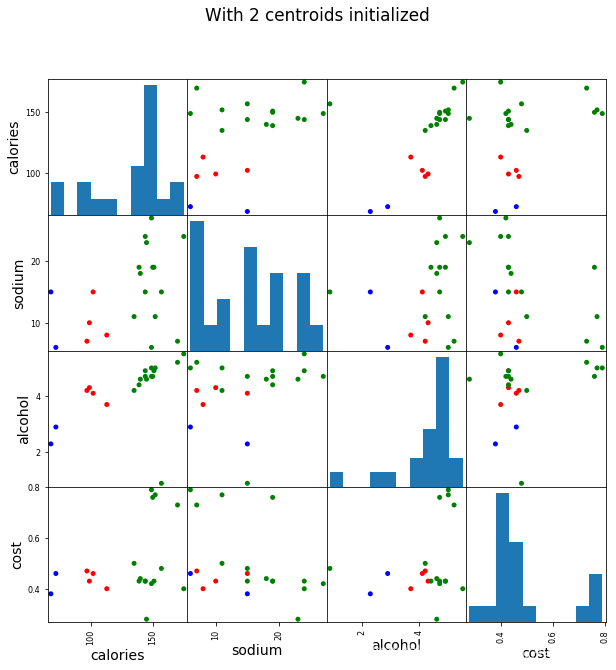
# 3簇
scatter_matrix(beer[["calories","sodium","alcohol","cost"]],s=100, alpha=1, c=colors[beer["cluster"]], figsize=(10,10))
plt.suptitle("With 3 centroids initialized")
使用 sklearn 的 StandardScaler 进行数据标准化操作
# 标准化
from sklearn.preprocessing import StandardScaler
# 实例化
scaler = StandardScaler()
# 执行
X_scaled = scaler.fit_transform(X)
X_scaled
array([[ 0.38791334, 0.00779468, 0.43380786, -0.45682969],
[ 0.6250656 , 0.63136906, 0.62241997, -0.45682969],
[ 0.82833896, 0.00779468, -3.14982226, -0.10269815],
[ 1.26876459, -1.23935408, 0.90533814, 1.66795955],
[ 0.65894449, -0.6157797 , 0.71672602, 1.95126478],
[ 0.42179223, 1.25494344, 0.3395018 , -1.5192243 ],
[ 1.43815906, 1.41083704, 1.1882563 , -0.66930861],
[ 0.55730781, 1.87851782, 0.43380786, -0.52765599],
[-1.1366369 , -0.7716733 , 0.05658363, -0.45682969],
[-0.66233238, -1.08346049, -0.5092527 , -0.66930861],
[ 0.25239776, 0.47547547, 0.3395018 , -0.38600338],
[-1.03500022, 0.00779468, -0.13202848, -0.24435076],
[ 0.08300329, -0.6157797 , -0.03772242, 0.03895447],
[ 0.59118671, 0.63136906, 0.43380786, 1.88043848],
[ 0.55730781, -1.39524768, 0.71672602, 2.0929174 ],
[-2.18688263, 0.00779468, -1.82953748, -0.81096123],
[ 0.21851887, 0.63136906, 0.15088969, -0.45682969],
[ 0.38791334, 1.41083704, 0.62241997, -0.45682969],
[-2.05136705, -1.39524768, -1.26370115, -0.24435076],
[-1.20439469, -1.23935408, -0.03772242, -0.17352445]])
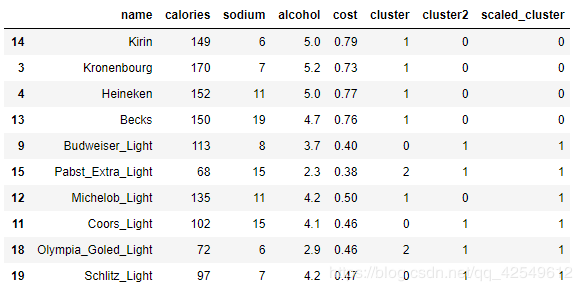
做完数据标准化后,再进行 K-eams 聚类
km = KMeans(n_clusters=3).fit(X_scaled)
beer["scaled_cluster"] = km.labels_
beer.sort_values("scaled_cluster").head(10)
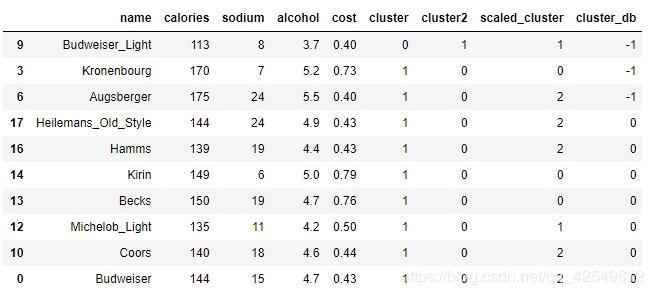
from sklearn.cluster import DBSCAN
# 实例化、执行
db = DBSCAN(eps=10, min_samples=2).fit(X)
# 得到聚类结果
labels = db.labels_
# 并入数据
beer['cluster_db'] = labels
beer.sort_values('cluster_db').head(10)
查看各个特征之间的聚类效果
pd.scatter_matrix(X, c=colors[beer.cluster_db], figsize=(10,10), s=100)
s(i)=b(i)−a(i)max{a(i),b(i)}s(i)=\frac{b(i)-a(i)}{max\{a(i),b(i)\}}s(i)=max{a(i),b(i)}b(i)−a(i)
s(i)={1−a(i)b(i),a(i)<b(i)0,a(i)=b(i)b(i)a(i)−1,a(i)>b(i) s(i)= \left\{\begin{matrix} 1-\frac{a(i)}{b(i)}, & a(i)b(i) \end{matrix}\right. s(i)=⎩⎪⎨⎪⎧1−b(i)a(i),0,a(i)b(i)−1,a(i)b(i)
计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, …, bik}
Si接近1,则说明样本i聚类合理
Si接近-1,则说明样本i更应该分类到另外的簇
若Si 近似为0,则说明样本i在两个簇的边界上
K-means聚类算法结果评估from sklearn import metrics
# 计算得分值(标准化数据)
score_scaled = metrics.silhouette_score(X,beer.scaled_cluster)
# 计算得分值(未做标准化数据)
score = metrics.silhouette_score(X,beer.cluster)
# 打印比较
print(score_scaled, score)
0.179780680894 0.673177504646
看起来聚类效果并不是很好,而且做完标准化后的数据在聚类任务上表现得并不好,原因可能就是在原始数据中不同的特征对聚类的影响度不同,在标准化后打破了各个特征的权重,所以在对数据预处理上,要根据数据的实际情况再进行操作
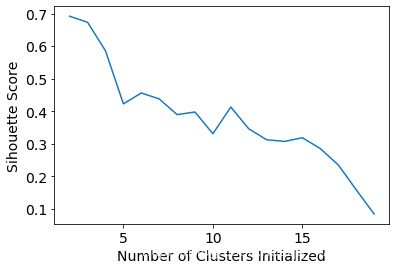
查看一下不同的 K 系数对聚类效果的影响:
# 算轮廓系数得分
scores = []
for k in range(2,20):
labels = KMeans(n_clusters=k).fit(X).labels_
score = metrics.silhouette_score(X, labels)
scores.append(score)
scores
[0.69176560340794857,
0.67317750464557957,
0.58570407211277953,
0.42254873351720201,
0.4559182167013377,
0.43776116697963124,
0.38946337473125997,
0.39746405172426014,
0.33061511213823314,
0.34131096180393328,
0.34597752371272478,
0.31221439248428434,
0.30707782144770296,
0.31834561839139497,
0.28495140011748982,
0.23498077333071996,
0.15880910174962809,
0.084230513801511767]
对输出的得分进行可视化展示
plt.plot(list(range(2,20)), scores)
plt.xlabel("Number of Clusters Initialized")
plt.ylabel("Sihouette Score")
# 算轮廓系数得分
labels_ = DBSCAN(eps=10, min_samples=2).fit(X).labels_
score = metrics.silhouette_score(X,labels_)
print(score)
0.49530955296776086
作者:六之