机器学习之PCA
PCA(主成分分析法—Principal Component Analysis)一. 求数据的前n个主成分二. 高维数据映射为低维数据
一. 求数据的前n个主成分
作者:皮蛋瘦肉粥_lzh
紧接着上次汇报的内容:
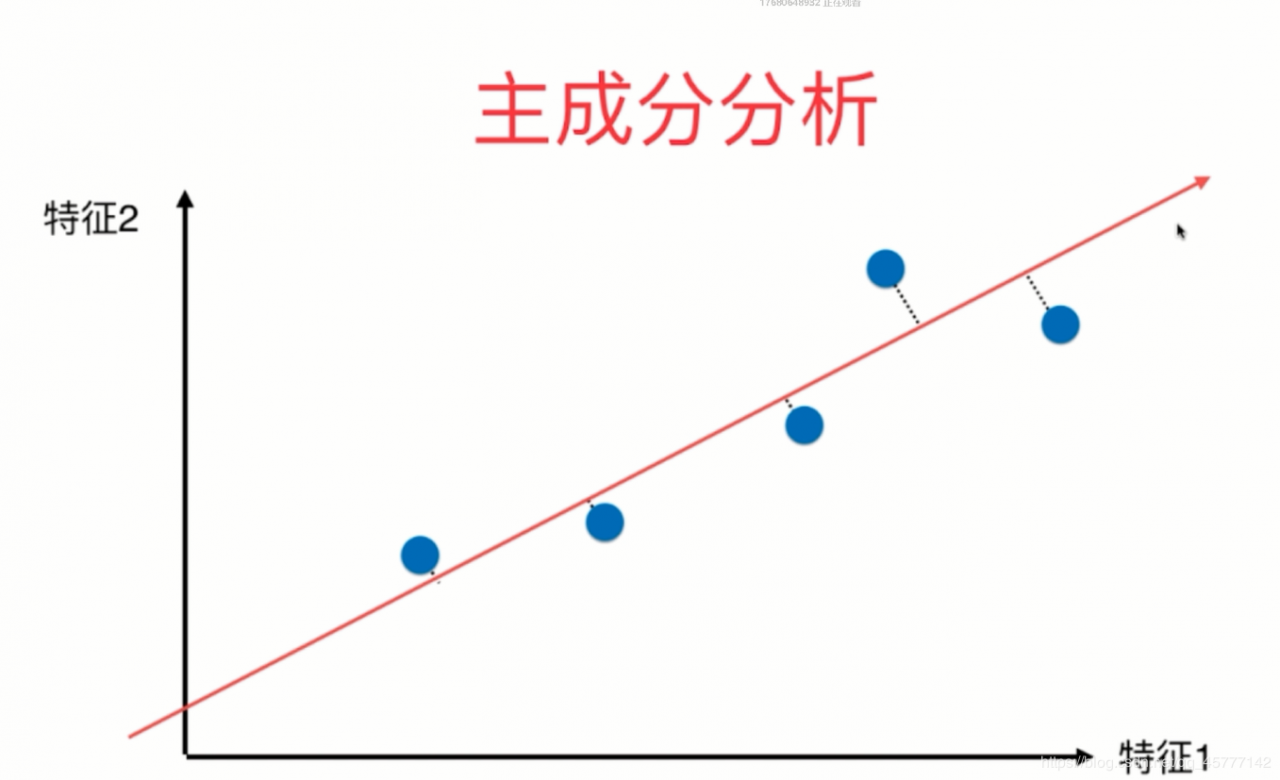
⟶将周围离散的点分别映射到这条直线上\stackrel{将周围离散的点分别映射到这条直线上}{\longrightarrow}⟶将周围离散的点分别映射到这条直线上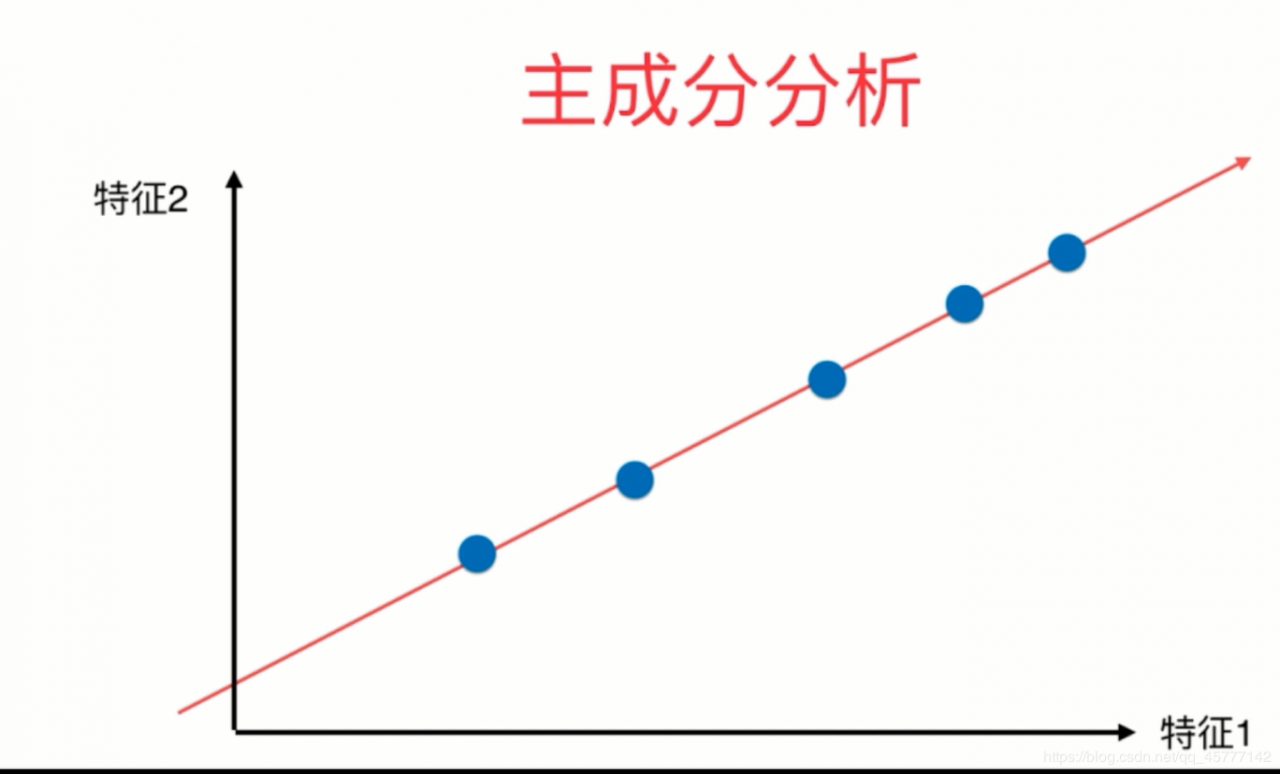
该直线所在的轴为我们的第一主成分,得到的结果就是这些样本点直接距离的方差是最大的
显然我们现在所求的不仅仅是局限在二维空间中,当维数增加时,我们如何求得下一个主成分?
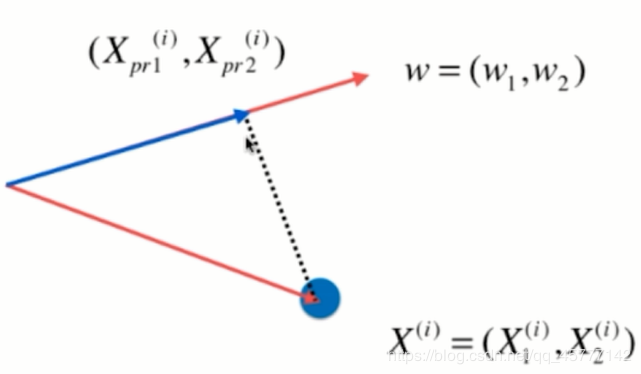 |
如左图所示,原来求直线向量:X(i)⋅w=∥X∥project(i)X^{(i)}\cdot w=\left \| X\right \|_{project}^{(i)}X(i)⋅w=∥X∥project(i) Xproject(i)=∥Xproject(i)∥⋅WX^{(i)}_{project} = \left \| X^{(i)}_{project}\right \|\cdot WXproject(i)=∥∥∥Xproject(i)∥∥∥⋅W |
|---|---|
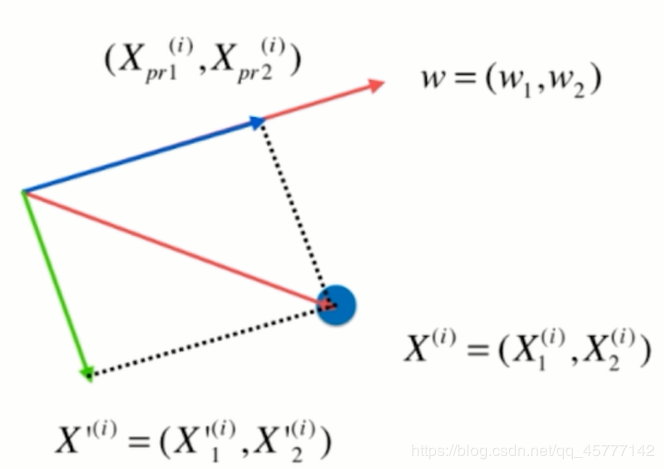 |
新求得的分量为(即图中绿色向量):X′(i)=X(i)−Xproject(i)X^{'(i)} = X^{(i)} - X^{(i)}_{project}X′(i)=X(i)−Xproject(i) |
其实道理是一样的:数据需要进行改变,将其在第一主成分上的分量去掉即可,原理为上方第二个图。
下面我们来编程实现这个前N个主成分的代码:
import numpy as np
import matplotlib.pyplot as plt #导入我们所需要的包
X=np.empty((100,2)) #随机创建一个我们需要的二维数组
X[:,0] = np.random.uniform(0.,100.,size=100)
X[:,1] = 0.75 * X[:,0] + 3. + np.random.normal(0.,10.,size=100)
def demean(X): #将X数据均值归一化
return X-np.mean(X,axis= 0)
X_demean = demean(X)
def f(w,X): #下面的步骤与之前的求简单二维PCA一模一样
return np.sum((X.dot(w)**2))/len(X)
def df(w,v):
return X.T.dot(X.dot(w))*2. / len(X)
def direction(w):
return w / np.linalg.norm(w)
def first_component(X,initial_w,eta,n_iters = 1e4,epsilon=1e-8): #梯度上升法
w = direction(initial_w)
cur_iter = 0
while cur_iter < n_iters:
gradient = df(w,X)
last_w = w
w = w + eta *gradient
w = direction(w)
if (abs(f(w,X) - f(last_w,X)) < epsilon):
break
cur_iter += 1
return w
initial_w = np.random.random(X.shape[1])
eta = 0.001
result=first_component(df_math,X_demean,initial_w,eta)
print(result) #此后我们设法减去第一主成分上的分量
X2 = np.empty(X.shape)
for i in range(len(X)):
X2[i] = X[i] - X[i].dot(w) * w
w2 = first_component(X2,initial_w, eta)
print(w2) #此时输出的值如果和原来W相乘 结果为0(因为2个垂直向量的乘积为0)
print(w.dot(w2)) #若结果为0 则该算法没毛病
#接下来我们尝试可以自己定义N的个数的N维度主成分分析法
def first_n_component(n , X, eta = 0.01, n_iters = 1e4, epsilon=1e-8):
X_pca = X.copy()
X_pca = demean(X_pca)
res = []
for i in range(n):
initial_w = np.random.random(X_pca.shape[1])
w = first_component(X_pca, initial_w, eta)
res.append(w)
X_pca = X_pca - X_pca.dot(w).reshape(-1,1) * w
return res
#我们还是以二维的数据来验证一下
print(first_n_component(2, X))
二. 高维数据映射为低维数据
上述所求虽然实现了多维数据主成分轴所在向量,但是数据仍然是高维的,如何对数据进行降维处理?
我们先从矩阵入手:
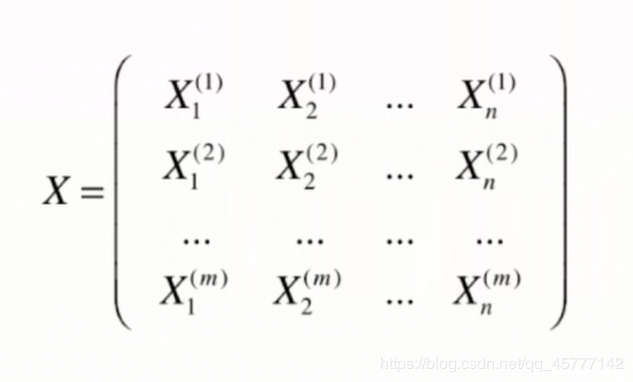 |
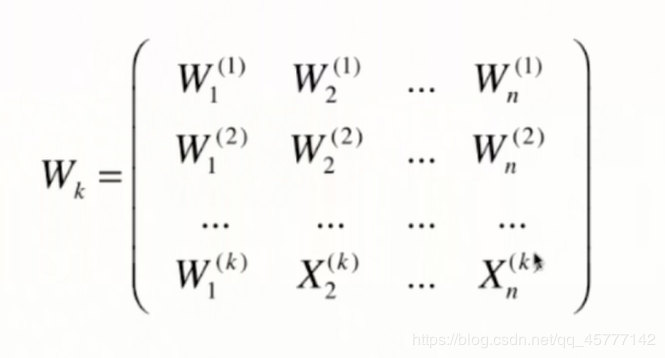 |
|---|---|
| 假设 X 数据有m个样本,n个特征 | 此时 W 为针对 X 求出来的前K个主成分 |
如何将样本数据 X 从 n 维转换为 k 维?
想象一下,如果我们将 X 中的一个样本和 k个w 分别作点乘的话,那么我们的要的数据便是这个一个样本在k个方向上对应的每一个方向大小。正常情况下 k < n,说这样我们便完成了 n 维样本 到 k 维的映射。矩阵相乘表示为:
X⋅WkT=XmX\cdot W^{T}_{k} = X_{m}X⋅WkT=Xm
下面我们实现PCA算法的源码:
import numpy as np
class PCA:
def __init__(self, n_components):
"""初始化PCA"""
assert n_components >= 1, "n_components must be valid"
self.n_components = n_components
self.components_ = None
def fit(self, X, eta=0.01, n_iters=1e4):
"""获得数据集X的前n个主成分"""
assert self.n_components <= X.shape[1], \
"n_components must not be greater than the feature number of X"
def demean(X):
return X - np.mean(X, axis=0)
def f(w, X):
return np.sum((X.dot(w) ** 2)) / len(X)
def df(w, X):
return X.T.dot(X.dot(w)) * 2. / len(X)
def direction(w):
return w / np.linalg.norm(w)
def first_component(X, initial_w, eta=0.01, n_iters=1e4, epsilon=1e-8):
w = direction(initial_w)
cur_iter = 0
while cur_iter < n_iters:
gradient = df(w, X)
last_w = w
w = w + eta * gradient
w = direction(w)
if (abs(f(w, X) - f(last_w, X)) < epsilon):
break
cur_iter += 1
return w
X_pca = demean(X)
self.components_ = np.empty(shape=(self.n_components, X.shape[1]))
for i in range(self.n_components):
initial_w = np.random.random(X_pca.shape[1])
w = first_component(X_pca, initial_w, eta, n_iters)
self.components_[i,:] = w
X_pca = X_pca - X_pca.dot(w).reshape(-1, 1) * w
return self
def transform(self, X):
"""将给定的X,映射到各个主成分分量中"""
assert X.shape[1] == self.components_.shape[1]
return X.dot(self.components_.T)
def inverse_transform(self, X):
"""将给定的X,反向映射回原来的特征空间"""
assert X.shape[1] == self.components_.shape[0]
return X.dot(self.components_)
def __repr__(self):
return "PCA(n_components=%d)" % self.n_components
紧接着我们自己通过测试来更感性的认识一下这个算法的强大:
小伙伴一定要记得把上述的Python代码(打包成PCA.py)和下面这个测试的放在一个目录下!!
import numpy as np
import matplotlib.pyplot as plt #导入我们所需要的包
X=np.empty((100,2)) #随机创建一个我们需要的二维数组
X[:,0] = np.random.uniform(0.,100.,size=100)
X[:,1] = 0.75 * X[:,0] + 3. + np.random.normal(0.,10.,size=100)
from PCA import PCA
pca = PCA(n_components= 1)
pca.fit(X)
print(pca.components_)
'''此处得到的是我们所求的主成分的2个方向'''
X_reduction = pca.transform(X)
print(X_reduction.shape)
'''此时样本的2个特征已经降为一个特征,同样也可以将一维的恢复为原先的二维'''
X_restore = pca.inverse_transform(X_reduction)
print(X_restore.shape)
'''最后我们来可视化一下'''
plt.scatter(X[:,0],X[:,1],color = 'b',alpha = 0.5)
plt.scatter(X_restore[:,0],X_restore[:,1],color = 'r',alpha = 0.5)
plt.show()
可视化结果为:
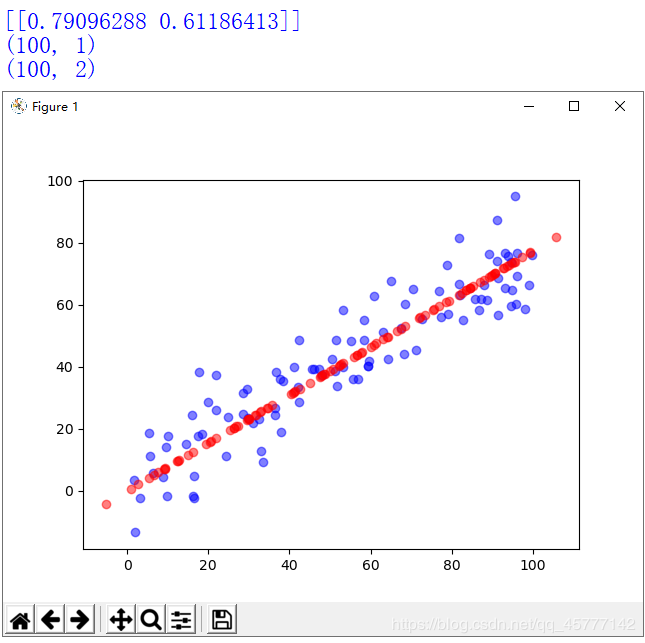
大家可以很清晰的看到 紫色的离散点为原来二维数组中X的点 ,而红色的点映射成一条直线便是我们所求的降维后的主成分!!
作者:皮蛋瘦肉粥_lzh
相关文章
Quirita
2021-04-07
Iris
2021-08-03
Kande
2023-05-13
Ula
2023-05-13
Jacinda
2023-05-13
Winona
2023-05-13
Fawn
2023-05-13
Echo
2023-05-13
Maha
2023-05-13
Kande
2023-05-15
Viridis
2023-05-17
Pandora
2023-07-07
Tallulah
2023-07-17
Janna
2023-07-20
Ophelia
2023-07-20
Natalia
2023-07-20
Irma
2023-07-20